Thème de recherche n°1 : Les ARN hélicases
ARN hélicase est un terme générique désignant un ensemble de familles de protéines appartenant aux superfamilles (SF) de protéines NTP-dépendantes. Elles sont impliquées dans la maturation de l’ARN, la réplication, la réparation de l’ADN, l’expression et le remodelage des complexes d’acides nucléiques. On connait au moins 7 de ces superfamilles qui utilisent soit l’ARN soit l’ADN comme substrat. Elles sont caractérisées par des motifs de liaison aux nucléotides qui sont très conservés. Les “coeurs catalytiques” de ces protéines sont constitués de feuillets beta parallèles entourés d’hélices. On les nomme domaine de type RecA car cette structure est similaire à celle de la protéine RecA, qui joue un rôle dans la recombinaison homologue chez E. coli. La majorité des protéines de ces superfamilles contiennent un seul domaine de type RecA, tandis que les familles de type SF1 et SF2 ont deux domaines de ce type reliés par un "linker". La vaste majorité des hélicases à ARN appartient à la famille SF2, mais quelques unes d’entre elles font toutefois partie de la famille SF1.
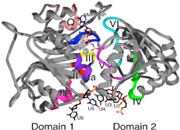
Fig. 1. Modélisation de la structure de l’hélicase à boite DEAD de levure, Ded1p, avec ses deux domaines de type RecA (résidus 117-529), réalisée à partir de la structure de Vasa, une protéine très proche de Ded1 (coordonnées pdb 2db3, Sendogu et al, 2006, Cell 125:287-300). Les ligands (AMP-PNP, Mg2+ et ARN U2-U6) ont été positionnés comme dans Vasa.
Les ARN hélicases utilisent l’énergie libérée par la liaison et par l’hydrolyse des nucléotides tri-phosphates. La plupart d’entre elles utilisent l’ATP ou le dATP comme source d’énergie, mais certaines peuvent utiliser l’UTP, le CTP ou le GTP. Même si on les appelle des “hélicases”, elles peuvent avoir bien d’autres activités. Par exemple, la protéine RIG-1 (retinoic acid-inducible protein) et la protéine MDA5 (melanoma differentiation-associated protein 5) reconnaissent l’ARN double brin produit lors des infections virales et peuvent activer les réseaux de signalisation antivirale. De même, la protéine SecA est un cofacteur du complexe bactérien SecYEG qui est impliqué dans la translocation des polypeptides à travers la membrane. D’autres ARN hélicases fonctionnent comme ARN chaperones capables de faciliter la formation d’ARNs fonctionnels, ou comme RNPases capables de dissocier les protéines liées à l’ARN, ou encore comme protéines d’assemblage capables de faciliter la formation de complexes ribonucléoprotéiques fonctionnels. Enfin, il a été montré que certaines hélicases peuvent dérouler des duplexes ARN-ARN ou ARN-ADN.
ARN hélicase: Pourquoi sont-elles importantes?
Les hélicases à ARN existent dans toutes les formes de vie, des bactéries aux eucaryotes supérieurs. Même les virus, comme HCV, codent pour leurs propres ARN hélicases, même s'ils utilisent également les hélicases de leurs hôtes. Les ARN hélicases sont associées à tous les processus impliquant l’ARN: transcription, épissage, transport, contrôle qualité, traduction et dégradation. De nombreuses hélicases sont essentielles et elles sont rarement interchangeables, ce qui indique leur fort degré de spécialisation dans les cellules. De ce fait, on les retrouve impliquées dans de nombreux processus biologiques, tels que le cycle cellulaire, le développement, le cancer, le vieillissement, les désordres neurologiques et d’autres maladies. Ce sont donc des cibles potentielles qui pourraient être utilisées comme agents prophylactiques et thérapeutiques.
ARN hélicase: ARN hélices: Qui sont elles?
Les ARN hélicases à boite DEAD ne peuvent lier l’ARN qu'en présence d’ATP, et sont capables d’hydrolyser l’ATP qu'en présence d’ARN. Elles constituent la plus grande famille d’ARN hélicases, et appartiennent à la famille SF2 qu’on appelle souvent la super-famille des protéines à boite DExD/H. On les retrouve pratiquement chez tous les organismes: la levure Saccharomyces cerevisiae en possède 25 et on en trouve 5 chez la bactérie Escherichia coli. Chez la levure, la plupart de ces protéines sont essentielles et non interchangeables. Chez E. coli, elles ne sont pas essentielles individuellement dans les conditions de culture de laboratoire. Elles sont impliquées dans tous les processus indiqués ci-dessus. Ce sont en général d’assez mauvaises hélicases in vitro, qui ne sont capables de dérouler que des duplexes d’ARN de relativement petites tailles (une douzaine de paires de bases). De plus, ces protéines ne présentent aucune directionalité ni processivité in vitro. On pense que ce sont leurs partenaires protéiques qui leurs confèrent directionalité et processivité in vivo. Les protéines à boite DEAD sont caractérisées par la présence de 9 motifs conservés qui jouent un rôle dans la liaison avec les ligands et qui leurs confèrent leur activité enzymatique. Leur nom vient des acides aminés caractéristiques du motif II (Asp-Glu-Ala-Asp), soit D-E-A-D. Cependant, il existe des protéines appartenant à la famille des hélicases à boite DEAD qui ont une séquence différente pour le motif II, et des protéines qui ont un vrai motif II mais qui ne font pas partie de cette famille. Les hélicases à boite DEAD sont caractérisées par d’autres marques, en particulier par le motif “Q” que nous avons découvert et caractérisé.

Fig. 2: Représentation schématique du “coeur” des protéines à boite D-E-A-D, montrant les 9 motifs conservés. Les positions des résidus qui interagissent avec les deux ligands, l’ARN et l’ATP (d’après la comparaison de plusieurs structures indépendantes) sont également représentées. Les flèches indiquent que le “coeur” se prolonge souvent par des extensions N- et C- terminales. Le “P” souligné indique une interaction avec un phosphate.
